背景
Crispr/Cas9在微生物中起到的作用就是识别入侵、将病毒的DNA录入自己的基因组,当病毒再次入侵时候,切割病毒DNA、RNA进而消除病毒的感染。CRISPR(Clustered regularly interspaced short palindromic repeats)是生命进化历史上,细菌和病毒进行斗争产生的免疫武器,简单说就是病毒能把自己的基因整合到细菌,利用细菌的细胞工具为自己的基因复制服务,细菌为了将病毒的外来入侵基因清除,进化出CRISPR系统,利用这个细菌免疫系统,细菌地把病毒基因从自己的染色体上切除,这是细菌特有的免疫系统。
原理
结构
Crispr在编辑基因的时候,需要2个成分①Cas9核酸酶;②gRNA(全称是guide RNA,引导RNA)(需要设计里面的部分序列)。
Cas9
其中Cas9核酸酶的基因已经被整合到商业化载体中,因此使用者只要自行设计sgRNA,然后构建Cas9/sgRNA表达载体,转染细胞,就可以行使基因组的定点基因编辑任务。
sgRNA结构
gRNA(guided RNA)是CRISPR基因敲除敲入系统中重要的组成部分,由两部分组成,分别是tracRNA和crRNA。其中crRNA引导cas9定位到要编辑的DNA序列附近,它有一段序列与tracrRNA的一部分是同源的,因此这两者可以部分结合。
tracrRNA的作用在于:形成一个茎环结构,与Cas9蛋白结合,示意图如下所示:

将上图的细节放大(图片来源于Sigma),如下所示:

如何发挥作用
Cas9和gRNA会形成一个Cas9核糖核蛋白(ribonucleoprotein,RNP)。这个RNP就能结合到基因组上的靶序列上。而基因组上的靶序列如果要被切割,需要满足两个条件:
第一,gRNA与基因组上有17-21个碱基的同源区,也被称为protospace。
第二,靶序列附近有PAM(protospacer adjacent motif),需要注意的是,在设计gRNA时,PAM并不是gRNA的一部分,找公司合成的gRNA时,里面是没有PAM序列的。
作用示意图如下所示:
在tracrRNA引导下,gRNA与基因组上的靶序列结合,进而RNP切割靶序列,形成一个DSB。
下图是Takara官网的原理示意图:

阶段 1:外源 DNA 采集
外源核苷酸是通过Cas 蛋白识别,入侵的短片段DNA(30-50个碱基对)被称为protospacers,作为间隔序列插入宿 主CRISPR 位点中,由重复序列隔开。对于I型和II型系统来说,protospacers来自入侵 DNA 中两侧出现2-5 个核酸结构(PAM,protospacer adjacent motif)的区域。一般 protospacers 连接在CRISPR 位点的一端,并且后者通过涉及 Cas1、Cas2 和 游离3’-hydroxyls 等元件的机制,牵引序列。Protospacer 的整合过程中也出现了牵引末端重新序列复制,这可能涉及宿主聚合酶和DNA修复机制。如下所示:
阶段 2:crRNA 合成
CRISPR RNA 在转录之后,生成初级转录产物:pre-crRNA,之后经过加工,又成为一组短小的 CRISPR衍生RNAs(crRNAs),这些crRNAs 每一个都包含有对应于之前遇到的外源DNA的对应序列。
阶段 3:靶向干扰
在pre-crRNA转录的同时,与其重复序列互补的反式激活tracrRNA(Trans-activating crRNA,tracrRNA)也转录出来,并且激发Cas9和双链RNA特异性RNase III核酸酶对pre-crRNA进行加工。加工成熟后,crRNA、tracrRNA和Cas9组成复合体,识别并结合于crRNA互补的序列,然后解开DNA双链,形成R-loop,使crRNA与互补链杂交,另一条链保持游离的单链状态,然后由Cas9中的HNH活性位点剪切crRNA的互补DNA链,RuvC活性位点剪切非互补链,最终引入DNA双链断裂(DSB)。CRISPR/Cas9的剪切位点位于crRNA互补序列下游邻近的PAM区(Protospacer Adjacent Motif)的5’-GG-N18-NGG-3’特征区域中的NGG位点,而这种特征的序列在每128bp的随机DNA序列中就重复出现一次。
补充:cas9解螺旋的过程
Cas9可以解开DNA,gRNA和target dsDNA配对是在解旋之后所进行(文献为:Sternberg, S. H., et al. (2014). “DNA interrogation by the CRISPR RNA-guided endonuclease Cas9.” Nature 507(7490): 62-67.)。步骤如下:
- gRNA和Cas9形成Cas9:gRNA complex.
- 这个complex来寻找双链DNA上的PAM位点。因为crisper/cas9源于细菌的防御机制,为了定点清除进入细胞内的“异己DNA而设计”,PAM就是evolutionary conserved关于“异己DNA”的特异标志,所以Cas9需要识别PAM也就不难解释了。现在分子生物学常用的Cas9源自S. Pyogenes这个细菌种属,这种Cas9的特异PAM是5’ NGG(N可以是任何nucleotide)。在人类基因组上GG占5%(每42bp就有一个),因此人的绝大多数基因都在PAM附近,也就意味着几乎所有人的基因都可以被Cas9识别。
- 当Cas9:gRNA complex识别到PAM的时候,Cas9就会把下游的双链DNA解旋,然后这个complex来根据gRNA的序列“扫描”解旋的DNA,一旦发现和gRNA序列互补的DNA,Cas9就会把DNA切开,完成操作。
下图是Doudna那篇paper的原理解释的figure

sgRNA的序列特征和设计
sgRNA的序列设计原理
gRNA 设计靶序列选择:在NCBI找到该基因CDS区,分析相应的基因组结构,明确CDS的外显子部分。按照基因本身的性质,选择候选的待敲除位点,确定待敲除位点。对于蛋白编码基因,如果该蛋白具重要结构功能域,可考虑将基因敲除位点设计在编码该结构域的外显子。可选择将待敲除位点放在起始密码子ATG后的外显子上。如果被敲除的靶标基因是microRNA或者lncRNA,可以将待敲除位点设计在编码成熟microRNA的外显子或在编码成熟microRNA的外显子的5’和3’侧翼序列。
gRNA设计:通常情况下我们仅需要在你的目的位置附近找到PAM序列:NGG(N代表任意核苷酸),然后找到其紧邻的上游20 nt即可 (如下图所示)。
从前面的原理中可以知道,这个gDNA是与基因组上的正义链是相同的。
目的位置的选择:我们绝大部分时候的目的是完全敲除一个基因,其原理是利用移码突变(CRISPR/Cas系统诱导产生indel)。
注:indel全称为insertion,deletion,即插入,缺失,缩写为indel,
插入如下:
生物原始基因序列:ATCGTCGAATCCGGTTG
发生突变基因序列:ATCGTCGAAatcgTCCGGTTG,其中的小写字母atcg为插入的序列,为插入突变;缺失:
生物原始基因序列:ATCGTCGAAtccgGTTG
发生突变基因序列:ATCGTCGAAGTTG,其中,原始序列里的小写字母tccg为缺失的序列,为缺失突变;
插入和缺失都相对于原始序列来说:缺失指,在原始序列中的某一段丢失;插入指,相对于原始序列在某个位置多了一段序列,即在这个位置插入了一段序列
因此我们一般选择距离起始密码子ATG下游200bp范围内的exon区域。另外,通常一个基因往往会转录成2个以上的转录本,所以请选择尽量靠近5’端的公共部分,保证每个转录本都会成功移码突变。
通常情况下,为了便于下游KO mutants的鉴定,我们往往可以作如下设计:即Cas9的切割位点刚好被一个酶切位点跨过,且附近至少100 bp内酶切位点唯一(下游的敲除突变也能通过测序的手段来鉴定)。
上图的酶切示意图就显示了,敲除突变无法被酶切,而野生型的则能够进行酶切。
不过现在已经有了很多在线的gRNA设计工具,现在就以其中的一个为例说明一下。
sgRNA的载体连接
现在已经有很多可以设计gRNA的工具,这里直接略过,直接看后文中所附的gRNA设计工具即可。
我们实验室自己使用的是U6-sgRNA-SFFV-spCas9-PURO,示意图如下所示:
 使用的酶切位点是
使用的酶切位点是BbsI,使用BbsI切开后的切口如下所示:

因此,在合成gRNA的两条链时,正义链上的oligo序列前面要加一个cacc,而负义链上的oligo序列前面要加上aaac。
例如我们最终确确定的gRNA序列为CTGTTTGTGCAGGGCTCCGA,它要在两边加上caccG,这个cacc与BsmBI酶切后的gtgg配对,加G是因为这个转录起始位点,必须要加上,如果本身第一个是G,则不需要添加,另一条是5’-TCGGAGCCCTGCACAAACAG-3’,它的两端要加上aaac与gttt配对。
那么最终需要合成的序列有2条:oligo1:5’-caccGCTGTTTGTGCAGGGCTCCGA -3’; oligo2:5’- aaacTCGGAGCCCTGCACAAACAGc-3’。加粗部分与经BbsI酶切后的载体互补配对,BbsI的酶切位点如下所示: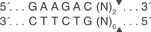
BbsI酶的信息如下所示:
全称:BbsI-HF
货号:R3539
保存条件:负20度
缓冲液:CutSmart Buffer(NEB带有HF(即High Fidelity)字样的酶都支持CutSmart)
cas9基因编辑案例分析
这个案例来源于Takara官网案例。
目标序列DNA的3'末端必须要有一个PAM(proto-spacer adjacent motif)序列,此序列为5'-NGG-3'。PAM序列上游的20个核苷酸就是我们要编辑的靶序列(也就是crRNA),Cas9核酸靶将会切除PAM上游的约3个核苷酸。
这里需要注意几点:
第一:寻找靶序列中的PAM序列,也就是NGG,我们需要注意的是,PAM序列并不是gRNA的一部分,因此在合成gRNA时,里面是没有PAM序列的;
第二:RNP在切割靶序列时,是可以切割任意一条链的DNA的(可能是正义链,也可能是反义链,总之在做敲除时,最终目的就是破坏DFNA)。

上图的流程是分三步:
- 确定靶序列的PAM序列,如上图中红色方框所示部分,是TGG;
- 在PAM上游数20个核苷酸;
- 合成gRNA(这个gRNA就是TGG前面的20个核苷酸序列)。
文献案例
再来看一个文献中的案例:
文献信息如下所示:
|
|
文献中是使用了cas9编辑了斑马鱼的etsrp基因,文献中的示意图如下所示:

红色方框画的就是靶序列,在Ensembl数据库中检索斑马鱼的etsrp外显子2序列,如下所示:
|
|
导入Snapgene,标记出来gRNA序列,如下所示:

从上图可以看到,这个gRNA序列就是位于正义链上。
实验流程
载体构建流程
1. Oligo退火形成duplex
| Component | Volume |
|---|---|
| oligo1 | 1ul(100uM) |
| oligo2 | 1uL(100uM) |
| H2O | 8uL |
| total | 10uL |
PCR仪 95℃ 5 min,缓慢降温至室温1h,1:200稀释duplex。
2. 质粒酶切
| Component | Volume |
|---|---|
| lentiCRISPR plasmid | 2ug |
| BbsI-HF(不同的质粒有不同的酶切位点) | 1uL |
| 10X buffer | 2uL |
| H2O | X uL |
| total | 10 uL |
加入以上成分,于37摄氏度酶切15分钟(不同的酶有不同的酶切时间,这个要看说明书)。此处使用的Cas9载体的gRNA插入位点如下所示:

3. 胶回收
大片段约11kb 回收,回收后的线性片段要放到负80度冰箱,因为BbsI这个酶消化后的质粒(我自己用的质粒)有一端是AAA,容易降解(如果没有,则放负20度即可)。
4. 连接
| Component | Volume |
|---|---|
| T4 DNA ligase(TaKaRa,2011A) | 1uL |
| 10 X DNA ligase buffer | 1uL |
| digested lentiCRISPR plasmid | 2uL |
| duplex | 6 uL |
| total | 10 uL |
室温连接4-6h,随后转化,转化时要把所有的连接体系加入到感受态中,因为载体的连接效率不高。
病毒包装体系
| Component | Volume |
|---|---|
| lentiCRISPR v2-gDNA plasmid | 4ug |
| psPAX2 packaging plasmid | 3ug |
| pMD2.G envelope plasmid | 1ug |
如果是使用Lipo3000进行病毒包括,则整个包括体系如下所示:
| 溶液1 | 溶液2 |
|---|---|
| ① OPTI-MEM 121.25μL | ① psPAX2(1.5μg= X μL)(X) |
| ② lipo3000 3.75μL(或7.5,第一次需要摸索条件) | ② pMD2G(1μg= YμL)(Y) |
| ③ 目的质粒(2.5μg= Z μL)(Z) | |
| ④ OPTI-MEM (125μL-X-Y-Z-10= μL) | |
| ⑤ P3000TM试剂 10μL(最后加) |
注:注:(1)包装质粒PAX2 :pMDG2:和目的基因质粒=3:2:5,因此前期工作,每种质粒的浓度最好为:[1]psPAX2(500ng/μL)[2]pMD2G(500ng/μL)[3]目的质粒(500ng/μL)
(2)Lip3000(μL)∶DNA(μg)的推荐比例为2.5∶1
转染
转染条件与常规的病毒转染相同。
转染48h后,加入puromycin来筛选细胞,1mL的培养基,加5μL的嘌呤酶素(1mg/mL),也就是说嘌呤霉素的终浓度是5μg/mL。
测序
加了嘌呤霉素的培养基要每天更换,每次里面都要含有5μg/mL的嘌呤霉素,这个时间要持续7天。在第7天的时候,取一定数量的细胞,提取DNA,测序,这一步的目的主要在于,确定是否存在有基因敲除的细胞,如果没有,就要重新包病毒进行敲除。
如果存在基因敲除的细胞,那么gRNA切割位点处就会产生套峰,表示有效编辑的效应为50%,如下所示:

单克隆的获取
当测序结果出现套峰时,表明这批细胞中存在被敲除的细胞,此时就可以分离单克隆了,分离单克隆主要有下面的几种方法。
有限稀释法
有有限稀释法的过程为:收集细胞,计数,将细胞的密度稀释为500个/mL,此时,用20μL移液枪吸取细胞悬液先点几个孔 ,每孔约2μL,然后放在倒置显微镜下观察 ,如每孔 1~2 个细胞,说明细胞浓度合适 ,则可一次点完 96 孔板,每个孔100uL。
这个过程也可以使用流式细胞仪分选,直接分到96孔板中,每孔种1个细胞。
直接挑单克隆
- 将细胞稀释到一定浓度(具体浓度自己摸索),直接种到10cm的培养皿中,这样的目的在于,每个细胞就会均值地铺到培养皿中,最终一个细胞就会长成一团细胞,这一团细胞就是单克隆;
- 生长一定时间后(例如7天),放到显微镜下观察,细胞数目足够了,用记号笔在培养皿下标记上;
- 吸掉培养基,用PBS清洗3次,吸取2uL的胰酶,滴到标记处,消化1分钟,然后再加10uL的培养基,此时用枪头把培养基吸走,细胞就进入了枪头,再把培养基打到24孔板中,让其生长。
单克隆的鉴定
第一种方法,根据设计,推荐使用酶切鉴定的方法(见设计部分),通量高;
第二种方法,就是提取细胞DNA,跑胶,在gRNA序列的上下游设计一对引物,产物长度约500bp,敲除的细胞的条带会缩减,由于gRNA只有20几个bp,因此普通的琼脂糖凝胶不容易看出差异,此时可以采用SDS凝胶,如果敲除的基因是非编码的序列,那么就采用测序法;
第三种方法,测序,如果不熟悉SDS凝胶,可以采用普通的琼脂糖凝胶,直接切胶,送测序;
第四种方法,如果有抗体,可以使用WB的方法。
gRNA设计网站收集
- ATUM公司:https://www.atum.bio/eCommerce/cas9/input
- flyCRISPR:http://tools.flycrispr.molbio.wisc.edu/targetFinder
- GeneArt CRISPR Search and Design Tool(ThermoFisher):
https://www.thermofisher.com/cn/zh/home/life-science/genome-editing/geneart-crispr/geneart-crispr-search-and-design-tool.html - GPP Web Portal:https://portals.broadinstitute.org/gpp/public/analysis-tools/sgrna-design
- CRISPOR:http://crispor.tefor.net/
参考资料
- CRISPR-Cas9全程实操教程:从gRNA设计到挑单克隆全程指导
- CRISPR是啥?有啥用呢?
- 一文掌握CRISPR/Cas9系统操作方法,超详细protocol来袭!
- CRISPR是什么鬼?
- 手把手教你搞定 CRISPR/Cas9 系统操作
- 一文掌握gRNA的设计,常用方法汇总解析
- 手把手教你利用CRISPR-Cas9系统精准敲除靶标基因
- 白友“冰糖”分享:CRISPR/Cas9系统操作实例
- CRISPR/Cas9系统操作实例分享之二:LentiCRISPR v2
- 手把手教你搞定 CRISPR/Cas9 系统操作
- NEB BbsI酶
- CRISPR-Cas9(三)—sgRNA设计好之后
- How to design sgRNA sequences